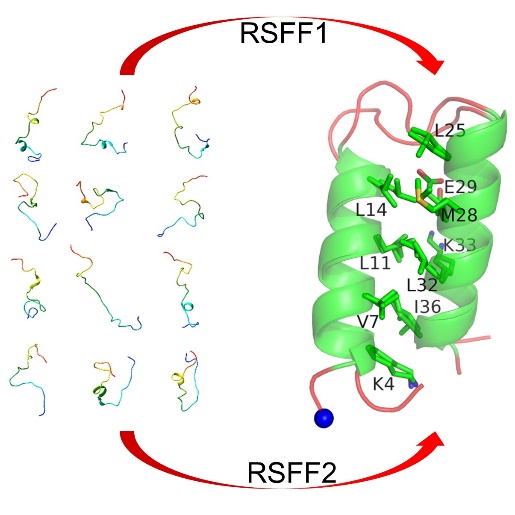
吴云东课题组在J. Phy. Chem. B.上发表论文报道全原子模型的alpha螺旋发卡的折叠模拟
Alpha螺旋发卡是一类非常重要的蛋白质结构域,能与DNA进行结合,行使重要的生物功能。Alpha螺旋发卡的构造非常简单,仅由两段相互作用的螺旋形成,两段螺旋中间由一个转角或者环连接。而且Alpha螺旋是单个alpha螺旋形成大型alpha螺旋结构域的中间体,是研究蛋白质折叠机制的关键模型。目前已有大量课题组运用各类实验方法致力于研究螺旋发卡的折叠机制,但是相互冲突的研究结论致使alpha螺旋发卡的折叠机制更加扑朔迷离。因此迫切需要进一步的研究,从原子的级别上揭示alpha螺旋发卡的折叠机制。在这个工作中,我们首次实现了全原子模型的alpha螺旋发卡的折叠模拟。模拟结果证明,不管是运用我们发展的RSFF1力场,还是RSFF2力场,动力学模拟都能给出与实验一致的三维结构,如下图所示
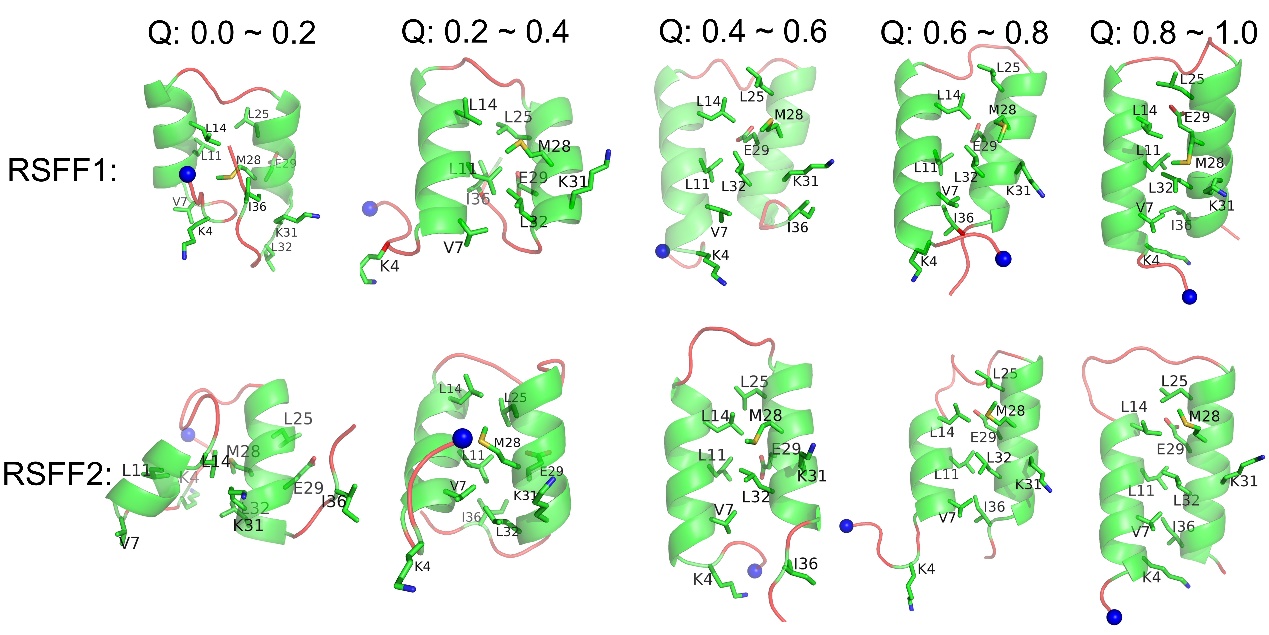
在研究过程中,我们分析了alpha螺旋发卡折叠过程中的自由能面,同时将Q值进行等间隔分区,分析局域RMSD和两段螺旋之间的关键相互作用,成功发现了alpha螺旋发卡的折叠机制。如下图所示,Q9-E15和E24-A30(靠近转角区域)分别是第一段螺旋和第二段螺旋中最稳定的区域,最早进行折叠,同时两段螺旋之间靠近转角区域的相互作用也开始形成。与此同时,位于两段螺旋中间的某些相互作用在前期就开始形成,如:L11和L32之间的相互作用,从而促使末端区域进行折叠。两段螺旋的N和C末端最后折叠,在整个折叠过程中,转角区域始终在2Å左右波动。针对以上观察到的折叠过程,我们提出了类似于拉链拉上过程的折叠机制。
该工作第一作者为我院博士后曾娟博士,发表于《物理化学杂志B》(Zeng, J.; Jiang, F.; Wu, Y.-D., Folding Simulations of an α-Helical Hairpin Motif αtα with Residue-Specific Force Fields. The Journal of Physical Chemistry B, DOI: 10.1021/acs.jpcb.5b09027)。
文章链接:http://pubs.acs.org/doi/abs/10.1021/acs.jpcb.5b09027
下一篇:Wu group published article on J. Phy. Chem. B. about Folding Simulations of an α-Helical Hairpin Motif αtα with Residue-Specific Force Fields.